Servicio de Microbiología
Prevención, diagnóstico, tratamiento y seguimiento de las enfermedades de origen infeccioso
Sistema Kiestra
El sistema Kiestra es una cadena automatizada para procesar muestras microbiológicas que nos permite, de una manera relativamente sencilla, dar salida a las más de 30.000 muestras de urinocultivo y frotis que se procesan anualmente.

Consta de 4 módulos:
- El sistema de almacenaje de medios de cultivo BD Kiestra™ SorterA™
- El sistema de identificación BD Kiestra™BarcodA™, donde las muestras se identifican con los medios de cultivo correspondiente según su procedencia clínica.
- El procesador de muestras BD Kiestra™ InoqulA™ automatiza el procesamiento de muestras tanto líquidas como no líquidas. Se basa en una tecnología de cuentas rodantes, que permite generar más colonias aisladas que el rayado basado en bucles (el que se realiza en una siembra manual) eliminando la necesidad de subcultivo, lo que puede acortar la identificación de patógenos y las pruebas de sensibilidad a los antimicrobianos.
- El incubador y capturador de imágenes BD Kiestra™ ReadA™ Compact donde los medios de cultivo ya sembrados se incuban. En este módulo también se realizan las capturas de imagen de los medios para poder realizar la lectura e interpretación del cultivo en la estación de trabajo.
Este innovador sistema de procesamiento de muestras microbiológicas ha permitido conseguir mejorar la productividad y la capacidad del personal mejorando el flujo de trabajo del laboratorio, así como la calidad de los resultados reduciendo el error humano y proporcionando resultados precisos y más rápidamente. Esto se ha conseguido optimizando el crecimiento bacteriano mediante tiempos de crecimiento y condiciones de incubación estandarizados en un entorno automatizado controlado.
Sistema MALDI-ToF
El desarrollo de la tecnología MALDI-ToF MS (matrix assisted laser desorption ionization-time of flight-mass spectrometry), en español: desorción/ionización láser asistida por una matriz con detección de masas por tiempo de vuelo, ha permitido la utilización de la espectrometría de masas en la identificación de microorganismos mediante el análisis de proteínas, principalmente ribosomales, a través de la creación de un espectro de masas que es específico para cada género y especie bacteriana.
Un espectrómetro de masas se compone de tres unidades funcionales, una fuente ionizante para transferir iones a las moléculas de la muestra en una fase gaseosa, un analizador de masas que separa los iones de acuerdo con su relación masa/carga y un dispositivo de detección para monitorizar los iones separados.

Una pequeña porción de una colonia de bacterias crecida en medio de cultivo sólido se deposita directamente sobre una placa metálica conductora. Posteriormente, a la placa con el microorganismo se adiciona una solución saturada de un compuesto orgánico de baja masa, denominada matriz.
Tras el secado, la muestra del microorganismo y la matriz se cristalizan y forman un depósito sólido de muestra incrustado en la matriz, la cual es esencial para la ionización. Se introduce en el espectrómetro de masas, en donde la mezcla es irradiada con pulsos cortos de un rayo láser. La interacción entre los fotones de las moléculas de láser y de la matriz, causados por la absorción de la energía del haz, desencadena una sublimación de la matriz en una fase gaseosa, seguida por la ionización de la muestra del microorganismo con mínima fragmentación.
Al ionizarse, las proteínas son aceleradas a través de un campo electrostático y luego son expulsadas en un tubo de vuelo al vacío donde se separan en función de su velocidad o tiempo de vuelo, llegando finalmente al detector de masas que genera información característica de la composición del microorganismo mediante un espectro de picos, frente a su relación masa/carga (m/z), llamada también huella digital de la masa de los péptidos (peptide mass fingerprinting).
Una vez generado, el perfil espectral del microorganismo de prueba es comparado automáticamente mediante un programa informático con una base de datos de espectros que es construida a partir de cepas de referencia, permitiendo la identificación del microorganismo, a nivel de especie.
Sistema Flow
Se emplea una plataforma semi-automatizada de PCR multiplex en tiempo real (sistema FLOW de Roche). El sistema permite la implementación de diversos paneles para la detección de múltiples patógenos gastrointestinales y respiratorios. El alicuotado de la muestra, extracción de ácidos nucleicos, preparación de las PCRs y detección en tiempo real se llevan a cabo en plataformas automatizadas que son controladas por un software específico. El sistema permite la implementación rápida de tests para la detección de patógenos emergentes, como ha sido el caso del SARS-CoV-2, dependiendo de las necesidades del laboratorio.

La trazabilidad de la muestra, intercambio de archivos entre los distintos equipos del sistema y la verificación de los resultados obtenidos son controlados por un software que está conectado al sistema informático de laboratorio (LIS).
Secuenciación masiva
Las técnicas de secuenciación masiva de nueva generación o next generation sequencing (NGS) nos permite secuenciar múltiples fragmentos de material genético de forma paralela, produciendo millones de secuencias al mismo tiempo.
Un experimento de secuenciación masiva consta de cuatro etapas principales: extracción del ADN de la muestra o aislado, preparación de las librerías, secuenciación propiamente dicha y análisis bioinformático e interpretación de los resultados.
En Microbiología, el uso de técnicas de NGS ha supuesto una revolución y tiene numerosas aplicaciones.

En nuestro laboratorio, utilizamos la secuenciación masiva en tres grandes campos. Por un lado, para el estudio de la microbiota: determinación de la composición (qué microorganismos tenemos en nuestra muestra) y la cantidad relativa de las comunidades microbianas (en qué proporción se encuentra cada uno de estos microorganismos). Este tipo de análisis se puede realizar de dos formas: amplificar un gen común a todos los microorganismos presentes en la muestra (targeted sequencing) o bien amplificar todos los genes presentes en la muestra (shotgun sequencing).
En el primer caso, se emplea el gen que codifica la subunidad 16S del ARN ribosomal (16S rARN) para bacterias. La segunda opción, en la que secuenciamos todos los genes presentes en la muestra, proporciona una cantidad de información mucho mayor que en el caso anterior y cada vez se está empleando con más frecuencia.
Por otro lado, las técnicas de NGS las estamos utilizando para secuenciar cepas bacterianas, lo que nos permite obtener el genoma completo y, de esta forma, identificar genes de resistencia y virulencia, comparación de cepas, entre otros aspectos. Es de gran utilidad en la identificación de los mecanismos de resistencia a antibióticos y en estudios de epidemiología molecular y dinámica de transmisión. Por otro lado, desde hace ya varios años, se emplea la NGS para la detección de mutaciones de resistencia a antirretrovirales en pacientes VIH.
IR Biotyper
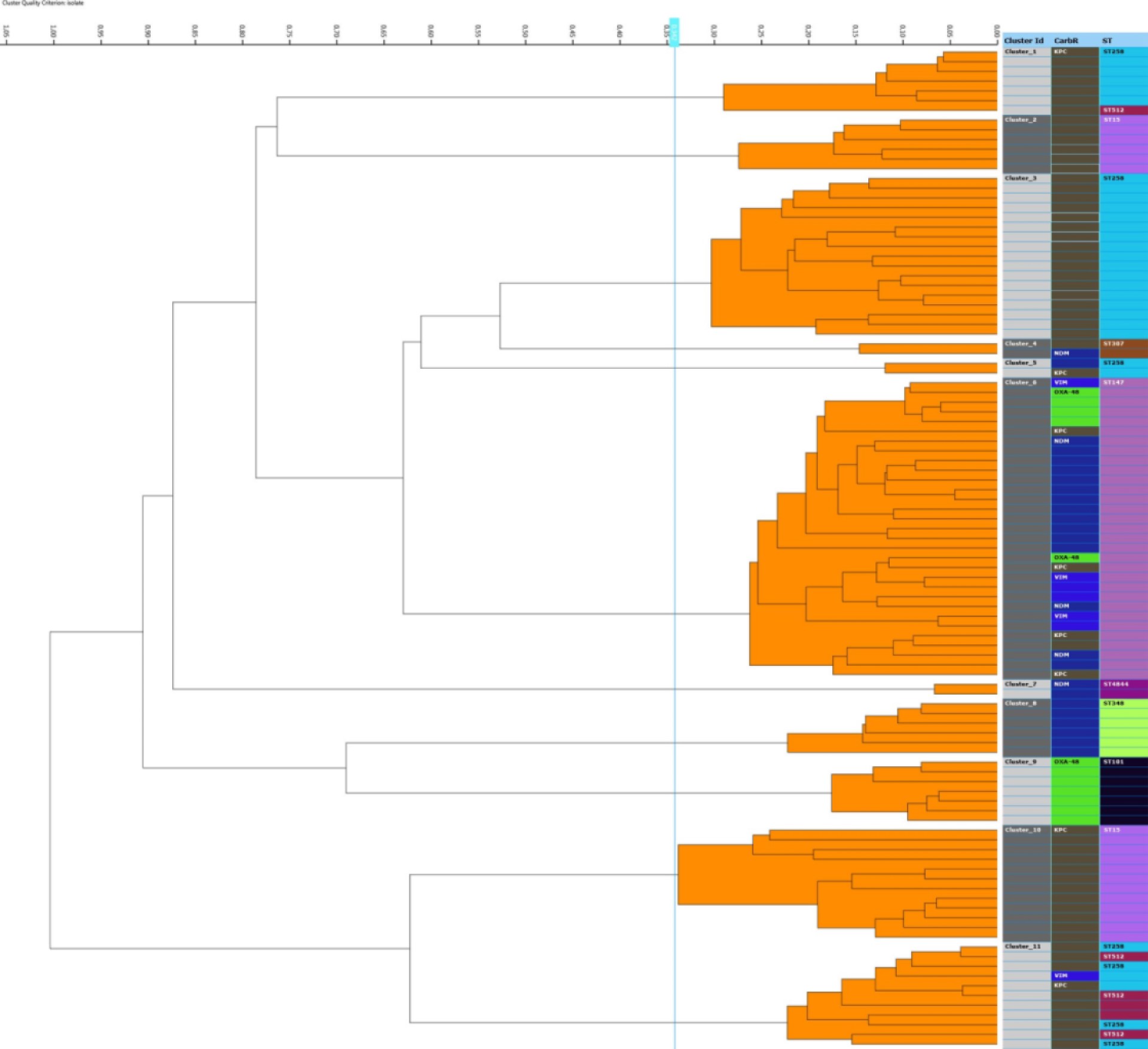
La espectrofotometría de infrarrojos por transformada de Fourier al análisis epidemiológico que realiza el IRBiotyper permite caracterizar la relación clonal de aislados bacterianos en base al análisis de las vibraciones de los carbohidratos que se encuentran en las glicoproteínas y la cápsula externa de la mayoría de bacterias.
El IRBiotyper obtiene un espectro de absorción de carbohidratos de superficies en un rango de longitud de onda (1.300-800 cm-1) que es característico para cada aislado y, de forma parecida al análisis por PFGE, los espectros se comparan mediante análisis informático para obtener una matriz de distancia que se utiliza para determinar el grado de relación fenotípica entre los aislados. Aquellos aislados que presenten un valor de distancia por debajo del valor de corte designado se consideran relacionados y se incluyen en el mismo cluster.
Presenta ventajas adicionales a técnicas tan ampliamente utilizadas como el PFGE y el MLST, ya que permite el análisis de un número muy superior de muestras en un tiempo mucho más corto lo que lo convierte en un método idóneo para la realización de estudios epidemiológicos en tiempo-real y la detección precoz de brotes nosocomiales por clones de alto riesgo.