
Bioquímica y Genética Molecular
Prevención, diagnóstico y control del tratamiento de numerosas enfermedades, mediante análisis bioquímicos y genéticos, empleando la tecnología más innovadora.
Genética Molecular
En la Sección de Genética Molecular se realizan las pruebas de laboratorio relacionadas con el diagnóstico genético de las enfermedades hereditarias. La actividad asistencial se desarrolla en torno a tres áreas: Genética y Citogenética Molecular, Citogenética Convencional y Consejo Genético.

El área de Genética y Citogenética Molecular es centro de referencia de la Red de Hospitales vinculados al Instituto Catalán de la Salud (ICS), ofreciendo pruebas de diagnóstico molecular para más de 100 enfermedades hereditarias y utilizando las técnicas más adecuadas en cada condición: PCR convencional, secuenciación Sanger, MLPA, Cariotipo Molecular a través de microarrays cromosómicos y secuenciación masiva-NGS (estudio por paneles o exoma).
La Sección participa en diferentes programas de diagnóstico/cribado de Cataluña, como son el cribado neonatal de la fibrosis quística, el programa de diagnóstico prenatal, que incluye el cribado de aneuploidías fetales en sangre materna (test prenatal no invasivo, NIPT), y el estudio germinal de cáncer hereditario en el Programa de Oncología de Precisión de CatSalut.
En el área de Citogenética Convencional, buena parte del trabajo está dedicada al diagnóstico prenatal, actuando como centro de referencia en Cataluña, utilizando tanto cariotipo convencional como hibridación fluorescente in situ (FISH).
En la consulta de Consejo Genético se proporciona diagnóstico clínico y asesoramiento genético y reproductivo en enfermedades de origen genético.
Jefa de Sección: Dra. Cèlia Badenas
Técnicas destacadas
Tecnología de arrays por Hibridación genómica comparada (aCGH)
La hibridación genómica comparativa o CGH (por sus siglas en inglés Comparative Genomic Hybridization) es un método de citogenética molecular empleado para analizar variaciones del número de copias (copy number variants o CNV). Relaciona el nivel de ploidía del ADN de una muestra en comparación con una muestra de referencia sin la necesidad de realizar un cultivo celular. El objetivo de esta técnica es comparar de manera rápida y eficiente dos muestras de ADN genómico para detectar ganancias o pérdidas de material genético.
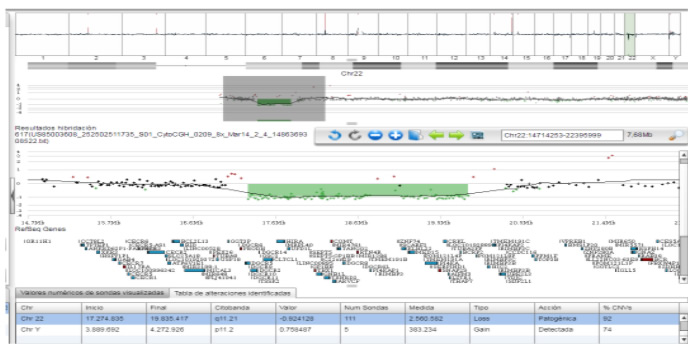
El aCGH empleado en nuestro laboratorio se compone de más de 60.000 sondas de tipo oligonucleótido. Este diseño permite interrogar a más de 300 regiones del genoma causantes de reordenamientos recurrentes relacionados con síndromes genómicos conocidos con una alta cobertura (en promedio 1 sonda/10Kb), ofreciendo un tiempo de respuesta en muestras prenatales de 10-15 días laborables desde la recepción de la muestra.
Test no invasivo prenatal en sangre materna (NIPT, Illumina, NGS)
El NIPT (del inglés, noninvasive prenatal testing) es una prueba de cribado para la detección de aneuploidías fetales en muestras de sangre materna de mujeres embarazadas (>10 semanas de gestación). Hasta hace poco, solo existían dos pruebas prenatales para la detección de las aneuploídias más frecuentes (trisomía 21, 13 y 18): el cribado bioquímico y la realización de una prueba invasiva (amniocentesis o biopsia de vellosidades coriónicas). Ambas pruebas presentan desventajas respecto a la opción NIPT. Los cribados bioquímicos ofrecen menor precisión y menor tasa de detección, y los procedimientos invasivos conllevan un riesgo de pérdida del embarazo. El uso del NIPT en el diagnóstico prenatal permite incrementar la tasa de detección de dichas aneuploidías (que puede llegar al ser del 99,7% para la trisomía 21) y reducir el número de procedimientos invasivos de confirmación (hasta un 89%).

El sistema utilizado para la detección prenatal de aneuploidias fetales mediante la secuenciación masiva del ADN fetal libre circulante en sangre materna es el VeriSeqp NIPT Solution v2 (Illumina). Este sistema es automatizado desde la preparación de la muestra a la secuenciación utilizando el secuenciador NextSeq550Dx, y permite la secuenciación simultánea de 24, 48 o 96 muestras. Los datos de secuenciación generados se analizan mediante un software de la propia casa comercial (Illumina). El riesgo de aneuploidías se valora mediante el cálculo del NCV (valor normalizado de cada cromosoma), que compara el número de lecturas de los cromosomas a valorar, en este caso los cromosomas 13, 18 y 21, respecto a unos cromosomas de referencia de la misma muestra. Un exceso en el número de lecturas indicará la presencia de una aneuploidía en el cromosoma determinado. Además, se incluye el cálculo de la fracción fetal (porcentaje de ADN de origen fetal). El ADN circulante es mayoritariamente de origen materno, y, en menor proporción, de origen fetal. Sin embargo el tamaño del ADN materno es ligeramente superior al del ADN fetal, por lo que el análisis del tamaño de los fragmentos secuenciados permitirá determinar el porcentaje de ADN fetal libre presente en la sangre materna. Como se ha comentado, el NIPT presenta un alto nivel de sensibilidad (>99% para T21 y >98% para T18 y T13) y especificidad (>99%), superiores a los del cribado sérico tradicional.
Secuenciación masiva del exoma completo
La secuenciación masiva del exoma completo permite obtener las variantes de todas las regiones génicas que codifican para proteínas. Aunque el exoma representa menos del 2% de todo el genoma (~45Mb), se estima que contiene el 85% de las variantes causantes de enfermedades mendelianas. Se trata de una tecnología de secuenciación de alto rendimiento.
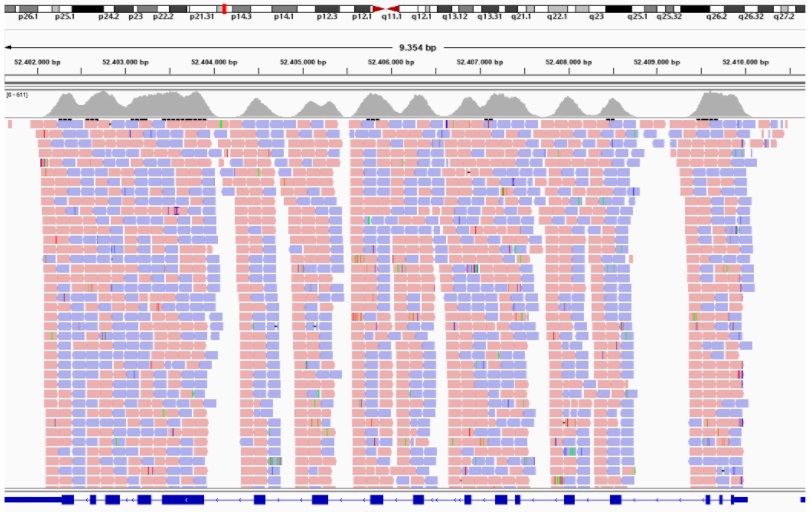
En nuestro hospital utilizamos el protocolo de secuenciación masiva Nextera flex for Enrichment con sondas Exoma completo (Illumina) y posterior secuenciación en la plataforma NextSeq550 (Illumina). Disponemos de un servicio de análisis bioinformático de los datos. Posteriormente un facultativo realiza el análisis genético. En base a la información clínica facilitada se filtra por paneles de genes definidos (por ejemplo hipertensión pulmonar, cardiomiopatías familiares o enfermedades metabólicas) o se crea un panel virtual específico (por ejemplo pacientes con discapacidad intelectual que presentan dismorfia u otros fenotipos asociados, o pacientes con enfermedades muy raras con una prevalencia < 1/1.000.000). Finalmente, se emite un informe con los resultados hallados, su interpretación y asesoramiento genético.
Los datos generados se almacenan y se ofrece la oportunidad de realizar ampliación de estudio en los casos que lo requieran (por ejemplo: nuevos genes identificados asociados a la enfermedad o aparición de otros fenotipos con origen genético). Una vez detectada la causa genética, se puede proceder al estudio de los familiares a riesgo y proporcionar un adecuado asesoramiento genético.
Investigación
“Nos mueve formar parte de un equipo multidisciplinar que une sus esfuerzos para mejorar las salud de las personas”
Líneas de investigación:
- Biología molecular de la reproducción y el desarrollo
- Melanoma: imagen, genética e inmunología
- Inflamación y enfermedad hepática
Proyectos en desarrollo:
- Decompensated cirrhosis: identification of new combinatorial therapies based on systems approaches
- Evaluación del efecto de la expansión del gen FMR1 sobre la reserva ovárica en mujeres jóvenes portadoras de la premutación
- Caracterización del gen REPS2 como nuevo gen candidato para los trastornos del neurodesarrollo
- FXPAC (Fragile X premutation associated conditions): caracterización clínica y análisis de la firma epigenética de metilación del ADN genómico
- Funció reproductiva masculina en pacients Covid-19
Acceso a la sección de Investigación del Hospital Clínic de Barcelona

